ÖZGÜN ARAŞTIRMA / ORIGINAL
ARTICLE
Doi: 10.5798/diclemedj.0921.2011.04.0053
İmmün sistemi baskılı
hastaların klinik örneklerinden soyutlanan Candida türlerinin PCR ile
belirlenmesi ve antifungal direnç genlerinin RFLP ve sekans analizi ile
saptanması
The identification of Candida species isolated
from clinical specimens of immunocompromised patients with PCR and
determination of antifungal resistance genes with RFLP and sequencing analysis
Serdar Susever, Yıldız Yeğenoğlu
İstanbul Tıp Fakültesi, Tıbbi Mikrobiyoloji Anabilim
Dalı, İstanbul, Türkiye
Yazışma Adresi / Correspondence: Dr. Serdar Susever, İstanbul Tıp Fakültesi, Tıbbi Mikrobiyoloji Anabilim
Dalı, İstanbul, Türkiye
Email: ssusever2@yahoo.com
Geliş Tarihi / Received: 12.04.2012, Kabul Tarihi / Accepted: 10.05.2012
ÖZET
Amaç: Bu çalışmanın amacı immun sistemi baskılı hastalardan
elde edilen klinik örneklerden izole edilen Candida türlerinde PCR tekniğinin
ve RFLP ve sekans analizi ile antifungal direnç genlerinin araştırılmasıdır.
Gereç ve yöntem: Çalışmada immün sistemi baskılı 200 hastadan alınan
klinik örnekler (96 bronkoalveoler lavaj, 56 biyopsi-apse, 8 kan, 15 periton
diyaliz sıvısı, 15 plevra sıvısı, 5 beyin omurilik ve 5 perikard sıvısı)
geleneksel ve moleküler yöntemler ile incelenmiş ve sonuçlar değerlendirilmiştir.
Örneklerden mantar DNAsı saptandıktan sonra elde edilen ürünün, multipleks PCR
yardımı ile tür düzeyinde tanısı yapılmış, antifungal direnci E-test, direnç
genleri ise RFLP analizi ile saptanmıştır.
Bulgular: İkiyüz örneğin 30unda (%15) kültür yöntemleri ile
pozitif sonuç alınmış [20 Candida
albicans (%67), beş Candida
parapsilosis (%17), beş Candida
tropicalis (%17)], 170 (%85) örnekte üreme olmamıştır. Genel primerler ile
yapılan PCR testi sonucunda kültürleri olumlu bulunan 30 örneğin tümünün mantar
DNAsı içerdiği belirlenmiştir. İzole edilen suşların biri E-test yöntemiyle
flukanozole dirençli, ikisi flukanozole doza bağlı duyarlı olarak saptanırken
27si duyarlı olarak bulunmuştur. Seçilen uygun primerler ile BamHI ve SalI
enzimleri kullanılarak RFLP uygulanmış, yapılan analizler sonucunda dirençli
bir C.albicans suşunda 600 bplik
bant görülmüştür. Candida dubliniensis
(950 bp) ve Candida krusei (360 bp)
ile yapılan multipleks PCR optimizasyonunda başarı
sağlanmıştır. Suşların ERG genine uygun primer çiftleri ile PCRları
yapıldıktan sonra yapılan sekans analizi sonucunda dirençli C.albicans suşu referans gen ile
karşılaştırıldığında D132E, E216D mutasyonları belirlenmiştir.
Sonuç: Moleküler test yöntemleri özellikle immün sistemi
baskılanmış hasta populasyonunun kısa sürede doğru tedavisine olanak
sağladıkları için yaşamsal açıdan önemlidir.
Anahtar sözcükler: antifungal direnç, Candida, E-test,
Erg11 proteini, kesilmiş parçaların uzunluk farklılığı (RFLP), polimeraz zincir
reaksiyonu (PCR),sekans analizi
ABSTRACT
Objectives: The aim of this study is to investigate PCR
technique and antifungal resistance genes with RFLP and sequencing analysis in
Candida species isolated from clinical specimens of immune-compromised
patients.
Materials and methods: Clinical samples (96 bronchoalveolar lavages,
56 biopsy-abscess, 8 blood specimens, 15 peritoneal fluid specimens, 15 pleural
fluid, 5 cerebrospinal fluid and 5 pericard fluid specimens) from 200
immunosuppressed patients were studied by conventional and molecular methods.
Antifungal susceptibility testing was performed by the E-test method. Firstly,
fungal DNA was isolated from specimens, and then the resultant products are
defined with multiplex PCR. Antifungal resistance and resistance genes were
established by E-test and RFLP analysis.
Results: Thirty of 200 samples (15%) were culture
positive [20 Candida albicans (67%),
five Candida parapsilosis (17%), five
Candida tropicalis (17%)], and 170 of
samples were found culture negative (85%). PCR with the universal primers
detected fungal DNA in all 30 culture positive samples. One strain was
determined as resistant; 2 strains were dose dependent susceptible and 27
strains were sensitive to fluconazole by E-test. The resistance gene (ERG11)
was detected by BamHI and SalI enzymes revealed fluconazole resistance in one
of C.albicans strains. The
identification was successful in Candida
dubliniensis (950 bp) and Candida
krusei (360 bp) with multiplex PCR. D132E and E216D mutations were detected
in sequencing of ERG 11 gene of this isolate and compared with reference gene
in GenBank by clustal analysis.
Conclusion: The molecular test methods supplies correct
therapy rather early in immunosuppressive patients therefore it is important
for the survival.
Keywords: Antifungal resistance, Candida, E-test, Erg11
protein, polymerase chain reaction (PCR), restriction fragment length
polymorphism (RFLP), sequencing analysis
GİRİŞ
Günümüzde giderek artış
gösteren immün sistemi baskılı hasta (İSBH) sayısı, beraberinde fırsatçı mantar
infeksiyonlarının da artışına olanak sağlamıştır. Başta Candida ve bunu
izleyerek Aspergillus cinsinden olmak üzere tüm fırsatçı mantarların son
yıllarda, özellikle terminal dönemlerinde olup farklı tanı almış immün baskılı
hasta gruplarından sıklıkla saptandığı görülmektedir. Öncelikli etken olan Candida albicansın hastane
infeksiyonlarında bakterilerden sonra ikinci sırada; mantarların kendi
içlerindeki sınıflandırmalarda ise ilk sırada yer aldığı birçok çalışma ile
gösterilmiştir. Candidalara bağlı kan infeksiyonları (kandidemi) oranı
1980lerde %2 iken, aynı yılın sonlarında %4e, 1990larda %8e yükselmiş,1999da
yapılan bir çalışmada ise bu oran %12 olarak bildirilmiştir. Kandidemilerden
kaynaklanan mortalitenin %50lerde bulunması, konunun ne denli önemli olduğunu
çarpıcı bir şekilde vurgulamaktadır.1,2
C.albicansın laboratuvar tanısında
ayırımda güçlük çekilen Candida
dubliniensis, kan infeksiyonlarından etken olarak sık saptanan Candida parapsilosis, flukonazole doğal
dirençli Candida krusei, flukonazole
giderek daha çok dirençliliği saptanan Candida
glabrata ve Candida tropicalisin
tür düzeyindeki çabuk tanılarının, tedavi ve dolayısı ile yaşamsal açıdan ne
denli önemli olduğu birçok çalışmada bildirilmiştir.3,4,5 Geleneksel
yöntemler ile izolasyon ve idantifikasyonun ortalama
bir haftayı kapsamasına karşın, moleküler tanı ile tüm bu uygulamalar için bir
buçuk günlük bir süre yeterli olmakta, antifungallere dirençlilik durumları da
aynı zamanda belirlenebilmektedir.
Antifungallere direnç konusu da günümüzde başlıbaşına önemli bir sorunu
teşkil etmektedir. Önceleri sınırlı sayıda olan antifungal
çeşitliliği son yıllarda artış göstermiş, aynı zamanda birçok nedene bağlı olan
antifungal dirençliliği de gündeme gelmiştir.6,7 Günümüzde özellikle
daha sık tercih edilen ve yan etkileri daha az bir azol bileşiği olan
flukonazole direnç konusu üzerinde önemle durulmakta ve konu ile ilgili olarak
çok sayıda çalışma sürdürülmektedir.7,8 Candida suşlarında
antifungal direncin moleküler mekanizmasının tanımlanmasında, genomik
çalışmalardan yararlanılmıştır. Çalışmalarda çoğunlukla ATP bağlayıcı
kaset ve esas kolaylaştırıcı super ailesi efluks pompa genleri ile ERG11 gibi
hedef enzimleri kodlayan genlerdeki mutasyonların gösterilmesi amaçlanmıştır.
Grp2p, Ifd1p, Ifd4p, Ifd5p, ERG10 genlerinin azol direnci ile ilişkisi
bulunduğu ve ERG10pnin ergosterol sentezinin ilk basamağında rol alan enzim
olduğu bildirilmiştir.9
Bu çalışmada polimeraz zincir reaksiyonunun ilk
basamağında genel primer çiftleri ile 550bpde klinik örnekteki mantar
varlığının saptanması, daha sonra türe özgü primerlerin kullanılması ile tür
düzeyinde tanısının yapılması amaçlanmış, bir diğer aşamada ise bu mantarların antifungallere
karşı direnç özellikleri ERG genlerine özgü primerler kullanılarak kesilmiş
parçaların uzunluk farklılığı (RFLP) analiz yöntemiyle araştırılmıştır. Direnç özelliği saptanan suşlarla, rastgele seçilen
duyarlı suşların PCR ürünleri purifiye edilip, sekans analizi yapılmış ve
çalışma sonunda RFLP yöntemi ile sekans sonuçları karşılaştırılarak
değerlendirilmiştir.
GEREÇ VE YÖNTEM
İstanbul Üniversitesi,
İstanbul Tıp Fakültesi, Tıbbi Mikrobiyoloji Anabilim Dalı, Mikoloji Bilim
Dalına, immün sistemi baskılı hastalardan alınarak gönderilen, 96
bronkoalveoler lavaj, 56 biyopsi-abse, sekiz kan, 15 periton diyaliz ve plevra
sıvısı, beş beyin-omurilik ve perikard sıvısı örneği geleneksel
(kültür-mikroskopi) ve moleküler yöntemler ile incelenerek değerlendirilmiştir.
İstanbul Tıp Fakültesi Mikroorganizma ve Kültür Koleksiyonları Merkezi
(KÜKENS)nden sağlanan, C.albicans
ATCC 10231 ve CBS 6431, C.parapsilosis
ATCC 22019, C.glabrata ATCC 15126, C.tropicalis KÜEN 1025, C.krusei ATCC 6258 ve C.dubliniensis CBS 7987 referans suşlar
olarak kullanılmıştır.
Geleneksel yöntemler: Sistemik mikoz kuşkulu örnekler üzerine %10-15lik
KOH+kalkoflor beyazı eklenerek hazırlanan preparasyon, floresan mikroskopta
incelenmiş ve mantar elemanları aranmıştır. Hasta örnekleri glikozlu Sabouraud
(pH: 5.5-6) ve beyin kalp infüzyon agar besiyerlerine
ekim yapılarak 25-30°Cde 30 gün süre ile inkübe edimiş; C.albicansın tanımlanması için tween 80li mısır unlu agara çizgi
ekim yapılarak klamidospor oluşumu araştırılmıştır. C.albicans dışındaki mayaların tür düzeyindeki tanıları
karbonhidratları asimile etme özelliğine dayanarak API
Klinik örneklerin ve referans suşların PCR ve multipleks PCR yöntemleriyle
tanımlanması: İlk olarak, DNA ekstraksiyon kiti [Modifiye Roche Kit Protokolü
(Roche, 11796828001)] yardımıyla referans suşlardan ve hasta örneklerinden
mantar DNAsı saptanmış ve elde edilen ürünlerin türe özgü primerler
kullanılarak multipleks PCR yöntemi ile tür düzeyinde tanısı yapılmıştır.
Primerlerin isimleri,dizilimleri,nükleotid sayıları (mer) ve çoğalttığı
uzunluklar Tablo-1de gösterilmiştir. Türe özgü multipleks PCR protokolünde;
altı tür için iki multipleks PCR paneli test edilmiştir. T-P panelindeki
primerler C.tropicalis, C.parapsilosisi (0.4 µl CTR 1 ve CTR2,
0.6 µl CPA1 ve CPA2), A-T panelindekiler ise C.albicans ve C.tropicalisi
(0.5µl CALB1, CALB2, CTR 1 ve CTR2) identifiye ederler (Tablo 1). Ana karışımı
sağlamak için 2 µl (~1 ng) dilüe genomik DNA, 20mM Tris-HCl (pH 8.4),
Antifungal duyarlılık: Klinik örneklerden izole edilen mayaların azol
dirençliliğinin belirlenmesinde E-test yöntemi (AB Biodisk, 1993)
kullanılmıştır. 24 saatlik Candida suşu 5 ml steril
RFLP: Antifungal direnç genlerinin belirlenmesinde RFLP yönteminden
yararlanılmıştır. Saflaştırma için elde edilen PCR ürününden 20 µl alınıp
üzerine, 2 ml
Sekans analizi: Direnç özelliği belirlenen suşlarla, rastgele seçilen
duyarlı suşların PCR ürünleri saflaştırılıp, gen düzeyinde farklılıklarının
saptanması için sekans analizi yapılmıştır (Sekans analizi Refgen firması
tarafından hizmet alımı olarak gerçekleştirilmiştir).
BULGULAR
Çalışma içeriğinde yer alan
200 immün sistemi baskılı hasta örneğinin 30unda (%15) kültür yöntemleri ile
olumlu sonuç alınmış [20 C.albicans
(%67), beş C.parapsilosis (%17), beş C.tropicalis (%17)], 170 (%85) örnekte
üreme olmamıştır.
İzole edilen Candida suşlarının, E-test yöntemiyle direnç özellikleri
araştırılmış; C.albicans suşlarından
birinin flukonazole dirençli, ikisinin doza bağlı duyarlı olduğu saptanmıştır.
27 Candida suşunun flukonazole duyarlı olduğu belirlenmiştir.
Genel primerler ile yapılan PCR testi sonucunda kültürleri olumlu bulunan
30 (%15) örneğin tümünün mantar DNAsı içerdiği belirlenmiştir. Mikroskopi,
kültür ve PCR olumlu on (%5); mikroskopi olumsuz kültür ve PCR olumlu 20 (%10)
örnek saptanmıştır. PCR olumlu kültür olumsuz örnek saptanmamıştır. Çalışmada
Kappa istatistiksel analiz testi yardımı ile kültür ve PCR uyumu 1.0
(p<0.001) olarak saptanmıştır. Uygulanan PCR yönteminin duyarlılığı ve
özgüllüğü %100 olarak belirlenmiştir.
Referans C.albicans, C.parapsilosis,
C.dubliniensis, C.tropicalis, C.kruseinin genel primer çiftleri ile
yapılan PCR sonuçları Resim 1de görülmektedir.
Çalışmamızda C.dubliniensis ve C.krusei ile yapılan PCR
optimizasyonunda başarı sağlanmıştır. Referans C.albicans ve C.dubliniensisin
türe özgü primerleri ile yapılan testler sonucunda C.albicansın iki ayrı referans suşunun (ATCC 10231 ve CBS 6431),
sırasıyla 273 ve 1050 bpde, C.dubliniensisin
(CBS 7987) 950 bpde (Resim 2), C.kruseinin
de 360 bpde (ATCC 6258) bant oluşturduğu saptanmıştır (Resim 3).
Referans C.parapsilosis ve C.tropicalisin genel ve türe özgü
primer çiftleri ile yapılan PCR analiz sonuçlarında, genel primer ile 550
bpde, türe özgü primerler ile 320 bpde (C.parapsilosis)
ve 357 bpde (C.tropicalis) bant
oluşturduğu görülmüştür (Resim 4).
Referans C.albicans, C.parapsilosis, C.tropicalisin multipleks optimizasyon çalışmaları sonucunda elde
edilen bantlar Resim 5te gösterilmiştir.
Klinik örneklerden saptanan C.albicans,
C.parapsilosis, C.tropicalisin türe özgü primerler ile yapılan multipleks PCR
sonuçları Resim 6da gösterilmiştir.
Seçilen uygun primerler ile BamHI ve Sal I enzimleri kullanılarak C.albicansa RFLP uygulanmış, yapılan
analizler sonucunda dirençli suşta 600 bplik bant saptanmıştır (Resim 7).
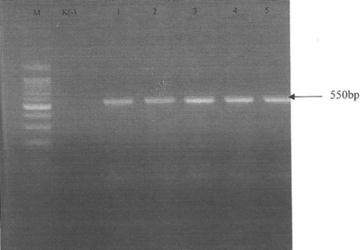
Resim 1. Referans Candida suşlarında
genel primer çiftleri ile PCR sonuçları. M: Moleküler standart (100 bp), sütun
1, 2, 3, 4, 5: Sırasıyla C.albicans
ATCC
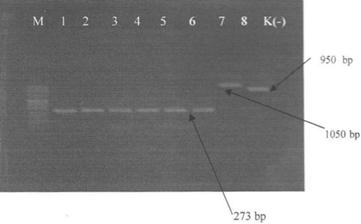
Resim 2. Referans C.albicans ve C.dubliniensisin
türe özgü primerler ile PCR sonuçları. M: Moleküler standart (100 bp), sütun 1,
2, 3, 4, 5, 6: C.albicans (ATCC
10231), sütun 7: Referans C.albicans
CBS 6431, sütun 8: Referans C.dubliniensis
CBS 7987, K(-): Negatif kontrol (H2O).

Resim 3. Referans C.kruseinin türe özgü primerler ile yapılan PCR sonuçları. M:
Moleküler standart (100 bp), sütun 1, 2: C.krusei,
K(-): Negatif kontrol (H2O).
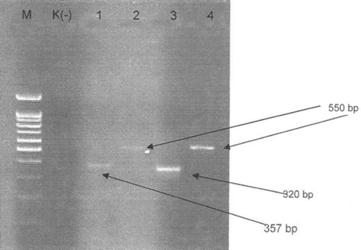
Resim 4. Referans C.tropicalis, C.parapsilosisin
genel ve türe özgü primer çiftleri ile PCR sonuçları. M: Moleküler standart
(100 bp), sütun 1: türe özgü primer C.tropicalis,
sütun 2 ve 4: Mantar DNAsı, sütun 3: C.parapsilosis,
K(-): Negatif kontrol (H2O).
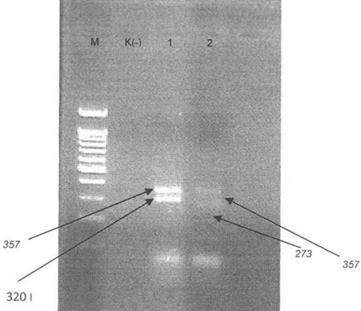
Resim 5. M: Moleküler standart (100
bp),K (-): Negatif kontrol (H2O), sutün 1: C.tropicalis, C.parapsilosis,
sütun 2: C.tropicalis, C.albicans.
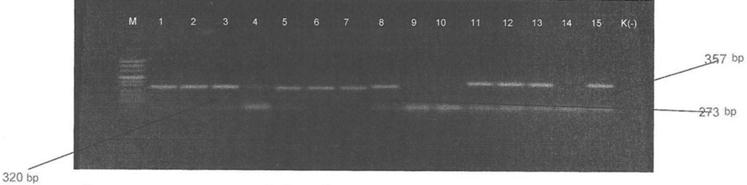
Resim 6. Klinik örneklerden saptanan C.albicans, C.parapsilosis, C.tropicalisin
türe özgü primerler ile multipleks PCR sonuçları. M: Moleküler standart (100
bp), K(-): Negatif kontrol (H2O), sütun 1-4, 8: C.parapsilosis, sütun 10,14: Negatif örnek, sütun 5-7: C.albicans, hat 11-13, 15: C.tropicalis.
Resim
Tablo1. Primer isimleri, dizilimi,
nükleotid sayısı (mer) ve çoğalttığı uzunluklar
|
Primer ismi |
Primer dizilimi (5 - 3) |
Nükleotid sayısı (mer) |
Çoğalttığı uzunluk |
|
Genel primerler |
~550 bp |
||
|
U1 |
GTG AAA TTG TTG AAA GGG AA |
20 |
|
|
U2 |
GAC TCC TTG GTC CGT GTT |
18 |
|
|
C.krusei |
360 bp |
||
|
CAK1 |
ACT ACA CTG CGT GAG CGG AA |
20 |
|
|
CAK 2 |
AAA AAG TCT AGT TCG CTC GG |
20 |
|
|
C.albicans |
~273 bp |
||
|
CALB1 |
TTT ATC AAC TTG TCA CAC CAG A |
22 |
|
|
CALB2 |
ATC CCG CCT TAC CAC TAC CG |
20 |
|
|
C.glabrata |
~423 bp |
||
|
CGL1 |
TTA TCA CAC GAC TCG ACA CT |
20 |
|
|
CGL2 |
CCC ACA TAC TGA TAT GGC CTA CAA |
||
|
C.parapsilosis |
~320 bp |
||
|
Grup I CPA1 |
TTG GTA GGC CTT CTA TAT GGG |
21 |
|
|
AllgrupCPA3 |
GCC AGA GAT TAA ACT CAA CCA A |
22 |
|
|
C.tropicalis |
~357 bp |
||
|
CTR1 |
CAA TCC TAC CGC CAG AGG TTA T |
22 |
|
|
CTR2 |
TGG CCA CTA GCA AAA TAA GCG T |
22 |
|
|
C.dubliniensis |
~950 bp |
||
|
CD1 |
GAA GGG CAT GCC TGT TTG AGA G |
22 |
|
|
CD2 |
ATC ACC TTC CCA CTA ACA CAT T |
22 |
|
|
ERG 11 direnç geni |
|||
|
CAF1 |
GAA AGG GAA TTC AAT CG |
17 |
750 bp |
|
CAR3 |
AAT ATA GTT GAG CAA ATG AAC G |
22 |
|
|
CAF4 |
GCT TCA AGA TCT TTA TTT GGT G |
22 |
1030 bp |
|
CAR6 |
AAC AAT CAG AAC ACT GAA TCG |
21 |
|
|
CPF1 |
ATT GCT AAC TTG TTG ATT GGT GTT |
24 |
600 bp |
|
CPR2 |
TGT AAT GGC ATG TGT AAT CTG AGG |
24 |
|
|
CTF1 |
TCT GAC ATG GTG TGT GTG TG |
20 |
500 bp |
|
CTR1 |
ATT GAT GCC ATC AAT GGC AG |
20 |
Elde edilen PCR ürünleri saflaştırılmış, gen düzeyinde farklılıklarının
belirlenmesi için sekans analizi uygulanmıştır. Azollere dirençli olarak
saptanan C.albicansın CAF1-CAR3
primerleri ile yapılan DNA dizi analizi sonuçları değerlendirildiğinde;
dirençli X13296 nolu referans geni ile AF153845 nolu duyarlı genin
nükleotidlerinin karşılaştırılması sonucunda farklı nükleotitlerin saptandığı
görülmüştür. Bu temele dayanarak RFLP ve E-test yöntemlerine göre dirençli
olarak belirlenen 102 nolu örneğin referans genlerden X13296 geni ile uyumlu
olduğu görülmüştür.153 ve 266 nolu aminoasitlerde mutasyon olduğu
saptanmıştır. Referans AF153845 nolu suşa ait duyarlı olarak bildirilen 153
nolu amino asitte E D ve 266ıncı amino asitte ise D E mutasyonu görülmüştür.
Dirençlilik saptanan 102 nolu C.albicansın,
dizisi yapılan ERG11 geninin 1528bplik tüm gen kısmı, NBCI gen bankasında
(http://www.ncbi.nlm.nih.gov) yayınlanmıştır (gen bankası no: GQ175788.1).
İstatiksel analiz: Çalışmada Kappa istatistiksel analiz testi yardımı ile
kültür ve PCR uyumu 1.0 (p<0.001) olarak saptanmıştır. Uygulanan PCR
yönteminin duyarlılığı ve özgüllüğü %100 olarak belirlenmiştir.12
TARTIŞMA
İnfeksiyon etkeni
olan/olabilen gerçek ve fırsatçı patojen mantarların tanımlanmasında, doğrudan
mikroskopik inceleme ve kültür günümüzde halen altın standart yöntemler olma
değerini korumaktadır. Ancak gerek zaman alıcı olması, gerekse her zaman olumlu
sonuç vermemeleri, bazı mantarlar açısından zor uygulanabilir ve yorumlanabilir
olması nedeniyle bu yöntemlerin yanı sıra; daha hızlı olan, duyarlılık ve
özgüllüğü yüksek yeni tanı yöntemlerine gereksinim duyulmuştur. Günümüzde
fenotipik tanı yöntemlerinde karşılaşılan sorunlar, moleküler yöntemlerin;
mikrobiyolojinin diğer alanlarında olduğu gibi gerektiğinde mikoloji alanında
da kullanılmasını zorunlu hale getirmiş, tanı, tedavi izlemi ve epidemiyoloji
konusunda giderek başarılı sonuçlar alınmasını sağlamıştır.
Watson-Crickin 1953de DNAnın yapısını tanımlamasından sonra, mantarları
ilgilendiren ilk genetik çalışmalar 1960 yılında Saccharomyces cerevisiaenin
genetik haritasının incelenmesiyle başlamıştır.13,14 Whelan, 1980de
C.albicans ile genetik çalışmalara
başlamış, Kurtz, 1986 yılında C.albicansa
ade2 geninin transformasyonunu bildirmiştir.2,7
1983 yılında Mullisin PCRı mikroorganizmaların moleküler tanılarında
kullanması sonucu mantarlar, bakteriler, virüs veya parazitlerin genetik
yapıları, konaktaki transkripsiyonel aktiviteler ve infeksiyona karşı oluşan
yanıtta; testin üstün analitik gücünden dolayı kısa zamanda başarılı sonuçlar
alınması, bu alanda yapılan çalışmalara hız ve değer kazandırmıştır.16
1992-1994 yılları arasında yapılan bir çalışmada, 601 kan örneği PCR
yöntemi ile incelenmiş; olumlu 109 örneğin 40ında C.albicans, 10unda C.tropicalis,
altışar tanesinde C.parapsilosis ve
Aspergillus flavus, 11inde C.glabrata,
sekizer tanesinde C.krusei ve Aspergillus fumigatus, ikisinde Candida guilliermondii, üçer tanesinde Candida kefyr ve Aspergillus versicolor, yedisinde Aspergillus niger, beşinde Aspergillus
nidulans saptanmıştır.17
BacT/Alert yöntemi ile incelenen 150 kan örneği kültürünün; 36sında C.albicans, 19unda C.glabrata, yedisinde C.parapsilosis,
beşinde C.tropicalis, üçünde C.krusei, ikisinde C.krusei+C.glabrata,
birinde C.glabrata +C.albicans, aynı sayıda örneğe uygulanan
PCR-EIA tekniği ile de; 35inde C.albicans,
18inde C.glabrata, yedisinde C.parapsilosis, beşinde C.tropicalis, üçer tanesinde C.krusei ve C.glabrata+C.albicans,
ikisinde C.krusei+ C.glabrata birlikte saptanmıştır.6
Nötropenik, ve hematolojik malignitesi olan 72 hastanın ateşli dönemlerinde
ve antifungal kullanımı sırasında alınan kan örnekleri PCR ve kültürel
yöntemler ile incelenmiş, PCR ile 31i (
Kültürü olumlu 234 kan örneğiyle yapılan çalışmada, 237 maya (
Antifungal tedavi gören dört hastaya ait örnekten kültürel yöntemler ile
olumsuz, PCR ile olumlu yanıt alındığı bildirilmiştir.18
Kan kültüründen multipleks PCR yöntemi ile yapılan çalışmada, iki çift
primer kullanarak C.glabrata (482 ve
483 bp), C.guilliermondii (248 bp), C.parapsilosis (229 bp), C.albicans (218 veya 219 ve 110 bp), C.tropicalis (218 bp), C.neoformans (201 bp) ve C.krusei (182 bp) tanımlanmış;
duyarlılığın %96.9, özgüllüğün ise %87.5 olduğu bildirilmiştir.3
Araştırıcılar tarafından multipleks PCR yöntemi ile iki ayrı panel altında
primer çifti kullanarak yapılan bir çalışmada A.fumigatus (385 bp), C.albicans (273 bp), C.glabrata (423 bp), C.parapsilosis (320 ve 300 bp), C.tropicalis (357 bp) ve C.neoformans (136 bp)de saptanmıştır.4
Moleküler yöntemlerin uygulandığı çalışmalardan anlaşıldığı gibi,
araştırıcılar mantara ait genetik materyeli saptamak için var olan çeşitli
yöntemlerden, kendi koşullarına uygun olanlarını yeğlemişlerdir. Bu çalışmada,
DNAnın saptanması amacı ile ticari DNA ekstraksiyon kiti kullanılmıştır.
Genel primerler ile yapılan PCR testi sonucunda kültürleri olumlu bulunan
30 (%15) örneğin tümünün mantar DNAsı içerdiği belirlenmiştir. Mikroskopi,
kültür ve PCR olumlu on (%5); mikroskopi olumsuz kültür ve PCR olumlu 20 (%10)
örnek saptanmıştır. PCR olumlu kültür olumsuz örnek saptanmamıştır.
Çalışmada, ITS1, ITS2, ITS3, ITS4, 5,8S rDNA ve 28S rDNA bölgelerinden
çoğaltılan genel ve türe özgü primerlerin kullanılması sonucunda mantarlara
özgü genel primer (550 bp) ve türe özgü primerlerin kullanılması ile: C.albicans (273 bp), C.parapsilosis (320 bp), C.tropicalis (357 bp), C.krusei (360 bp), C.dubliniensis (950 bp)de bant saptanmıştır. Bu sonuçlar
literatürle uygunluk göstermektedir.4,5
Bu çalışmada saptanmış olan %100 duyarlılık oranının Skladny ve ark.20
ve Hayette ve ark 21ın verileri ile tamamen; >%90 değer bildiren
Li ve ark.19 ve Chang ve ark.3ın sonuçları ile de yüksek
oranda benzer olduğu; özgüllük oranının (%100) ise diğer araştırıcılarca
bildirilen %96, %89 ve %87.5dan19,20,21 daha yüksek olduğu
görülmüştür.
Mantar infeksiyonlarının görülme sıklığının artması ve antifungallerin
yaygın kullanılması, tedavi sırasında dirençlerin görülmeye başlamasına neden
olmuştur. Direnç sorunu nedeni ile direnç mekanizmalarının araştırıldığı
çalışmalar günümüzde hız kazanmıştır. Antifungal direnç çok faktörlü
özelliklere bağlıdır. Direnç; in-vitro, moleküler ve klinik olarak üç başlık
altında toplanabilir. Antifungal dirençte olası mekanizmaları araştırmak için
mantarların tanısı ve identifikasyonunda kullanılan moleküler yöntemlerin
birçoğu kullanılmaktadır. Ayrıca antifungal ilaçlara direnç, duyarlılık ve
benzer suşlar arasında farklılığın tanımlanmasında da çeşitli moleküler
yöntemlerden (RFLP, karyotiplendirme, RAPD ve çeşitli PCR) yararlanılmaktadır.22
Azollerin hedefi olan 14 alfa demetilaz enzimini kodlayan gen ERG11dir.
Gelişen direnç mekanizmaları çoğunlukla bu enzimle ilgilidir. Mayalarda ERG11
olarak bilinen lanosterol 14 alfa demetilaz enzimi fungal canlılık için
gereklidir. Bu enzim lanosterolün ergosterole dönüşümünde lanosterol ve diğer
sterol prekürsörlerinden monooksidasyon tepkimesi ile 14 alfa metil grubunun
uzaklaştırılmasını sağlamaktadır. Enzimin flukonazol ile inhibisyonu, 14 alfa
metil gurubunun hidroksilasyonu için gereken oksijen aktivasyonunu
engellemektedir. Bunun sonucunda lanosterol ve 14 alfa metil steroidlerin
birikimi ve ergosterol tüketimi ile hücre ölümü gerçekleşir.23
Ayrıca hedef enzimin alfa heliks veya beta zincir gibi yapılarında oluşan
mutasyonlar nedeniyle meydana gelen yapısal değişiklikler sonucu bağlanmalar
etkilenerek afinite azalması ile direnç gelişmektedir. Bir çalışmada,
flukonazole direnç gelişiminde etkili olan ERG11 mutasyonlarının oranı %20
olarak bildirilmiştir.24
Çalışmamızda seçilen uygun primerler ile BamHI ve Sal I enzimleri
kullanılarak RFLP uygulanmış, yapılan analizler sonucunda dirençli bir suşta
600 bplik bant saptanırken, 29 suşta bant görülmemiştir.
Sonuç olarak, bu çalışmada geleneksel yöntemler ile identifikasyonu 4-5 gün
gibi bir süreyi kapsayan ve flukanazole intrensek direnç gösteren yaşamsal
önemi olan C.kruseinin, PCR yöntemi
uygulanarak, hızlı tanısı başarıyla gerçekleştirilmiştir. C.albicans gibi klamidospor oluşturan; asimilasyon fermentasyon
testleri ile kesin tanısı yapılamayan, ancak antifungal dirençliliği C.albicanstan daha fazla olduğu bilinen
C.dubliniensisin de PCR
optimizasyonu sağlanmış, türe özgü primerler kullanılarak bir tek PCR ile Candida
türlerinin identifikasyonunun yapılabileceği gösterilmiştir. RFLP ve E-test
yöntemlerine göre dirençli olarak saptanan 102 nolu örneğin referans genlerden
X13296 geni ile uyumlu olduğu saptanmıştır. Farklı görülen nükleotidlerin
aminoasitlere çevrildiğinde, 153 ve 266 nolu aminositlerinin farklı olduğu
saptanmıştır. Referans AF153845 nolu suşa ait duyarlı olarak bildirilen 153
nolu amino asitte E D ve 266ıncı amino asitte ise D E mutasyonu görülmüştür.
Saptanan mutasyonlardan, E153D; Marichal,25 D266E ise Löfflerin 26
yayınları ile uyum göstermektedir. Saptanan E153D mutasyonu birinci bölgede
olup substrat veya inhibitör giriş kanalına yakın olduğundan azollerin bağlanma
afinitelerini etkilemektedir Azollerin afinitesinde oluşan etkilenmenin; ya
ilacın girişini doğrudan etkileyerek ya da mutasyonlara bağlı helikal yapıda
oluşan değişiklikler nedeni ile olduğu bildirilmiştir.25
Doza bağlı duyarlı suşların saptanmasında; kullanılan E-testin yanısıra
sekans çalışmaları da yapılarak, amino asit mutasyonunun olup olmadığı
araştırılmıştır. Ülkemizde bu tip çalışmaların sürdürülmesi ve epidemiyolojik
olarak antifungal direnç genlerinin saptanması, antifungal direnç
mekanizmalarının aydınlatılmasını sağlayacak, özellikle ağır mikoz tablosu
gösteren immün sistemi baskılı hasta grubunun daha hızlı ve doğru tedavisinde
klinisyene de büyük ölçüde yardımcı olacaktır.
*Bu çalışma TÜBİTAK tarafından (Proje No:107S449) desteklenmiş ve 34. Türk
Mikrobiyoloji Kongresinde sunulmuştur, Poster No.166 (Kuzey Kıbrıs Türk
Cumhuriyeti, 7-11 Kasım 2010)
Teşekkür: Çalışmanın istatistiksel
değerlendirmesini yapan Doç. Dr. Halim İşsevere teşekkür ederiz.
KAYNAKLAR
1. Hazen KC, Howell SA. Candida, Cryptococcus,
and other yeasts of medical importance, Murray PR, Baron EJ, Jorgensen JH,
Pfaller MA, Yolken RH (eds): Manual of Cilinical Microbiology, 8th edn. ASM
Press,Washington, 2003:1693-710.
2. Kurtz MB, Cortelyou MW, Kirsch DR.
Integrative transformation of Candida
albicans, using a cloned Candida ADE2 gene. Mol Cell Biol 1986;6(1):1429.
3. Chang HC, Leaw SN, Huang AH, Wu TL, Chang TC.
Rapid identification of yeasts in positive blood cultures by a multiplex PCR
method. J Clin Microbiol 2001;39(10):3466-71.
4. Luo G, Mitchell TG. Rapid identification of
pathogenic fungi directly from cultures by using multiplex PCR., J Clin
Microbiol 2002;40(8):2860-5.
5. Romeo O, Racco C, Criseo G. Amplification of
the hyphal wall protein 1 gene to distinguish Candida albicans from Candida
dubliniensis. J Clin Microbiol 2006;44(7):2590-2.
6. Sanglard D, Bile J. Current understanding of
the modes of action and resistance mechanisms to conventional and emerging
antifungal agents for treatment of Candida of infections, Calderone RA (ed):
Candida and Candidiasis, 1th edn kitabında ASM Press, Washington, 2002:349-83.
7. Whelan WL, Partridge RM, Magee PT.
Heterozygosity and segregation in Candida
albicans. Mol Gen Genet 1980; 180(1):10713.
8. Sanglard D, Ischer F, Bille J. Role of
ATP-binding-cassette transporter genes in high-frequency acquisition of
resistance to azole antifungals in Candida
glabrata. Antimicrob Agents Chemother 2001;45(4):1174-83.
9. Arıkan S. Fungal
hastalıklarda genomik ve proteomiklerin yeri ve önemi. ANKEM Derg 2009;23(Ek
2): 53-6.
10. Clinical and Laboratory Standards Institute
(CLSI): Reference method for broth dilution antifungal susceptibility testing
of yeasts, Approved Standard 3rd edn, CLSI Document M27-A3, Clinical and
Laboratory Standards Institute,Wayne, PA 2008;28(14).
11. Xu Y, Chen L, Li C. Susceptibility of
clinical isolates of Candida species to fluconazole and detection of Candida albicans ERG11 mutations. J
Antimicrob Chemother 2008;61(4):798-804.
12. Alpar R. Spor Bilimlerinde
Uygulamalı İstatistik, 2. Baskı, s.219-55, Nobel Tıp Kitabevi, İstanbul (2001).
13. Günalp A. Gen ve Moleküler
Biyoloji, 1st edn, , Hacettepe Üniversitesi Yayınları, Ankara,
1965;1:226-63.
14. Hawthorne DC, Mortimer RK. Chromosome
mapping in Saccharomyces centromere-linked genes. Genetics 1960;45(8):1085-110.
15. Shin JH, Nolte FS, Morrison CJ. Rapid
identification of Candida species in blood cultures by a clinically useful PCR
method. J Clin Microbiol 1997;35(6):1454-9.
16. Ausubel FM, Brent R, Kingston RE et al.
Short Protocols in Molecular Biology, 3rd edn, John Wiley&Sons, New York,
1995:642-50.
17. Einsele H, Hebart H, Roller G, Loffler J,
Rothenhofer I, Muller CA. Detection and identification of fungal pathogens in
blood by using molecular probes. J Clin Microbiol 1997;35(6):1353-60.
18. Maaroufi Y, Heymans C, De Bruyne JM,
Duchateau V, Rodriguez-Villalobos H. Rapid detection of Candida albicans in clinical blood samples by using a TaqMan-based
PCR assay. J Clin Microbiol 2003;41(7):3293-8.
19. Li YL, Leaw SN, Chen JH, Chang HC, Chang TC.
Rapid identification of yeasts commonly found in positive blood cultures by
amplification of the internal transcribed spacer regions 1 and 2. Eur J Clin
Microbiol Infect Dis 2003;22(11):693-6.
20. Skladny H, Buchheidt D, Baust C, Krieg-Schneider
F, Seifarth W, Leib-Mosch C. Specific detection of Aspergillus species in blood
and bronchoalveolar lavage samples of immunocompromised patients by two-step
PCR., J Clin Microbiol 1999;37(12):3865-71.
21. Hayette MP, Vaira D, Susin F, Boland P, Christiaens
G, Melin P. Detection of Aspergillus species DNA by PCR in bronchoalveolar
lavage fluid. J Clin Microbiol 2001;39(6):2338-40.
22. Whıte TC, Marr KA, Bowden RA. Clinical,
cellular, and molecular factors that contribute to antifungal drug resistance.
Clin Microbiol Rev 1998;11(2):382-402 .
23. Haitao Ji, Zhang W, Zhou Y, Zhang M, Zhu J,
Song Y. A three-dimensional model of lanosterol 14α-demethylase of Candida albicans and its interaction
with azole antifungals. J Med Chem 2000;43(13):2493-505.
24. Favre B, Didmon M, Ryder NS. Multiple amino
acid substitutions in lanosterol 14α-demethylase contribute to azole
resistance in Candida albicans.
Microbiology 1999;145(10):2715-25.
25. Marichal P, Koymans S, Willemsens S, Bellens
D, Verhasselt P, Luyten W, Borgers M,Ramaekers FC, Odds FC and Vanden Bossche
H. Contribution of mutations in the111cytochrome P450 14α-demethylase (Erg
11p, Cyp 51p) to azole resistance in Candida
albicans. Microbiology 1999;145: 2701-13.
26. Löffler J, Kelly SL, Hebart H, Schumacher U,
Lass- Flörl C, Einsele H. Molecular analysis of cyp 51 from fluconazole
resistant Candida albicans strains.
FEMS Microbiol Lett1997;151:263-8.